Ngs клиника: Клиника ЭКО в Москве — центр репродукции (репродуктивной медицины), вспомогательные репродуктивные технологии
Врачи
Специалисты высочайшего уровня с многолетним опытом медицинской практики
- 1.Весь персонал
- 2.Репродуктологи
- 3.Эмбриологи
- 4.Генетики
- 5.Урологи
- 6.Медсестры
- 7.Аптека
- 8.Руководство
- 9.Административный персонал
- 10.Гинекологи
- 11.Врачи УЗД
- 12.Эндокринологи
Корнилов Николай Валерьевич
Медицинский советник
Репродуктолог
Подробнее
Морев Владимир Владимирович
Главный врач. Врач уролог-андролог. Врач высшей категории.
Врач уролог-андролог. Врач высшей категории.
Подробнее
Забелкина Ольга Игоревна
Заведующая отделением ВРТ Репродуктолог
Подробнее
Кирсанов Андрей Адольфович
К.м.н, медицинский директор
Репродуктолог
Подробнее
Чаликова Марина Борисовна
Генеральный директор
Подробнее
Каменецкий Борис Александрович
К.м.н, репродуктолог
Подробнее
Лобзева Диана Андреевна
Заведующая международным отделом
Репродуктолог
Подробнее
Гвасалия Русудан Гивиевна
Репродуктолог
Подробнее
Федоренко Иллона Игоревна
Репродуктолог
Подробнее
Павлова Марина Николаевна
Заведующая эмбриологической лабораторией
Подробнее
Вислогузова Александра Владимировна
Репродуктолог
Подробнее
Осепаишвили Мака Николаевна
Репродуктолог
Подробнее
Курило Алексей Олегович
Репродуктолог
Ведущий специалист
Подробнее
Волкоморов
Виктор Владимирович
Лабораторный генетик
Подробнее
Кораблева
Надежда Владимировна
Акушер-гинеколог-репродуктолог
Подробнее
Мурза Галина Валерьевна
Эмбриолог
Подробнее
Тарасова Лидия Ивановна
Исполнительный директор
Подробнее
Фасахутдинова Лейсан Ханифовна
Биолог
Подробнее
Кухтина Юлия Андреевна
Врач клинической лабораторной диагностики
Подробнее
Вяткина Светлана Вячеславовна
Заведующая лабораторией генетики
Подробнее
Прокошева Алена Сергеевна
Подробнее
Стрижова Мария Алексеевна
Подробнее
Васильев Роман Вячеславович
Подробнее
Пустовалова
Наталья
Медсестра
Подробнее
Кречмар Марина Валерьевна
Заведующая отделением клинической генетики, врач-генетик
Подробнее
Синельникова Марина Сергеевна
Медсестра
Подробнее
Корнилова Екатерина Евгеньевна
Старшая медсестра
Подробнее
Печаткин
Никита Михайлович
Коммерческий директор
Подробнее
Тарасова Оксана Александровна
Помощник врача
Подробнее
Шестакова Карина Павловна
Менеджер донорского банка
Подробнее
Жукова
Наталья Геннадьевна
Директор по развитию азиатского направления
Подробнее
Латкова Ксения Сергеевна
Помощник врача
Подробнее
Калинина Екатерина Борисовна
Фармацевт
Подробнее
Михайлова Евгения Олеговна
Старший администратор
Подробнее
Павлов Александр Владимирович
Начальник ИТ-отдела
Подробнее
Обернихина
Екатерина Александровна
Лабораторный генетик
Подробнее
Андреева
Наталья Викторовна
Эмбриолог
Подробнее
Денисова
Валентина Михайловна
Репродуктолог
Подробнее
Романова
Марина Викторовна
Репродуктолог
Подробнее
Алиханашвили
Наталья Викторовна
Акушер-гинеколог
Подробнее
Смородина
Наталья Львовна
Акушер-гинеколог
Подробнее
Бодренкова
Дарья
Эмбриолог
Подробнее
Мунгалова
Анастасия Дмитриевна
Репродуктолог
Подробнее
Румянцева
Екатерина Валерьевна
Биолог
Подробнее
Любан
Анна Константиновна
Репродуктолог
Подробнее
Малкина
Анна Владимировна
Эмбриолог
Подробнее
Терехова
Наталия Андреевна
Биолог
Подробнее
Билибина
Анна Александровна
Эмбриолог
Подробнее
Долгая
Юлия Федоровна
Эмбриолог
Подробнее
Калиберова
Екатерина Евгеньевна
Медицинская сестра
Подробнее
Рыжова
Анна Алексеевна
Медицинская сестра
Подробнее
Кадонцева
Евгения Андреевна
Заведующая аптечным пунктом
Подробнее
Ворожцова
Татьяна Александровна
Провизор
Подробнее
Шупик
Ольга Игоревна
Медицинская сестра
Подробнее
Голтелова
Анжела Вячеславовна
Медицинская сестра
Подробнее
Боровик
Наталья Викторовна
Эндокринолог
Подробнее
Златина
Елена Александровна
Врач акушер-гинеколог
Подробнее
Демидова Надежда Капитоновна
Медицинская сестра
Подробнее
Беленкова Кристина Алексеевна
Медицинская сестра
Подробнее
Волошина Ольга Ивановна
Медицинская сестра
Подробнее
Новикова Анастасия Дмитриевна
Лабораторный генетик
Подробнее
Колмагорова Анастасия Егоровна
Менеджер по развитию
Подробнее
Мариничев Александр Владимирович
Менеджер международного направления
Подробнее
Лисина Эльвира Владимировна
Врач ультразвуковой диагностики
Подробнее
Кувшинова Лидия Александровна
Врач ультразвуковой диагностики
Подробнее
Кравчук Екатерина Никодимовна
Эндокринолог
Подробнее
Боровик Наталья Викторовна
Эндокринолог
Подробнее
Жолобова Ольга Олеговна
Заместитель генерального директора по развитию агентской сети
Клиника репродукции и генетики «NGC» — 28 врачей, 205 отзывов | Санкт-Петербург
Врачи клиники репродукции и генетики «NGC»
61 отзыв
Каменецкий Борис Александрович
Репродуктолог
Стаж 31 год
1 категория, кандидат наук
В принципе, на приеме у Каменецкого Бориса Александровича мне все понравилось. Дело в том, что прежде чем к нему записаться я читала много отзывов о нем в интернете, сложилось непонятное мнение, думал…
Дело в том, что прежде чем к нему записаться я читала много отзывов о нем в интернете, сложилось непонятное мнение, думал…
Расписание недоступно
16 отзывов
Романова Марина Викторовна
Репродуктолог, акушер, гинеколог
Стаж 27 лет
Спокойный, грамотный врач, профессионал своего дела! Обратилась к Романовой М.В. в ФБГУ им. Алмазова СПБ в конце 2016 г. для обследования и процедуры «Инсеминация». Врач все подробно объясняет, назна…
Расписание недоступно
36 отзывов
Забелкина Ольга Игоревна
Репродуктолог, акушер, гинеколог
Стаж 22 года
кандидат наук
Забелкина Ольга Игоревна – наш семейный ангел! Пять лет назад с первого протокола у нас всё получилось! Доченьке четыре года, и с врачом мы её ещё не знакомили. Решили с мужем, что отличный повод дл…
Решили с мужем, что отличный повод дл…
Расписание недоступно
20 отзывов
Кирсанов Андрей Адольфович
Репродуктолог, акушер, гинеколог
Стаж 37 лет
высшая категория, кандидат наук
Чудо-врач! Адекватный, все по делу, мало говорит, делает, что и как надо! Очень ему благодарна. Специалист с большой буквы!
Расписание недоступно
18 отзывов
Любан Анна Константиновна
Репродуктолог
Стаж 15 лет
Хочу начать сразу с главного! Благодаря Анне Константиновне, у меня через 15 лет бесплодия сейчас есть счастье, которому уже 2,5 месяца! 15 долгих лет мы с мужем жили в ожидании малышки. К своим 34 го…
Расписание недоступно
Показать всех врачей
Информация
График врачей
Медали
1
Отзывы
205
Цены
19
Фотографии
17
График работы врачей
Свободной записи по этой специальности нет.
Подробности уточняйте по телефону (812) 317-11-64
Медали
Врачи клиники заняли призовые места в премии ПроДокторов 2020
Отзывы
Пациент
+7-903-09XXXXX
14 сентября в 16:59
-1.0 плохо
Здание и помещения
Оборудование и медикаменты
Отношение медперсонала
Комфорт пребывания
Время ожидания
Нормально
Хорошо
Ужасно
Ужасно
Ужасно
Проверено (3)
Посетили в августе 2022
пр-кт Петровский, д. 2, стр. 3
Пациент
+7-951-67XXXXX
21 августа в 16:33
Проверено (2)
Посетили в марте 2021
пр-кт Петровский, д. 2, стр. 3
Пациент
+7-900-65XXXXX
14 августа в 14:52
+1. 6
отлично
6
отлично
Здание и помещения
Оборудование и медикаменты
Отношение медперсонала
Комфорт пребывания
Время ожидания
Отлично
Отлично
Нормально
Отлично
Отлично
Проверено (3)
Посетили в январе 2022
пр-кт Петровский, д. 2, стр. 3
Пациент
+7-950-04XXXXX
30 июля в 12:35
+1.6 отлично
Здание и помещения
Оборудование и медикаменты
Отношение медперсонала
Комфорт пребывания
Время ожидания
Отлично
Отлично
Отлично
Отлично
Нормально
Проверено (3)
Посетили в июле 2022
пр-кт Петровский, д. 2, стр. 3
Пациент
+7-952-29XXXXX
16 марта в 00:16
-2. 0
ужасно
0
ужасно
Здание и помещения
Оборудование и медикаменты
Отношение медперсонала
Комфорт пребывания
Время ожидания
Ужасно
Ужасно
Ужасно
Ужасно
Ужасно
Проверено (3)
Посетили в феврале 2022
пр-кт Петровский, д. 2, стр. 3
Цены
Гинекологические услуги
4200₽ — 25000₽
УЗИ
1800₽ — 2000₽
Андрологические услуги
4500₽ — 4500₽
ЭКО
8900₽ — 161600₽
Показать все цены
Фотографии клиники репродукции и генетики «NGC»
16 изображений
Видео клиники репродукции и генетики «NGC»
1 видео
Метод NGS в клинике ISIDA Киев — Клиника ISIDA Киев, Украина
Метод NGS увеличивает возможности ЭКО
В 2016 году, клиника ISIDA одной из первых в Украине внедрила метод NGS, позволяющий еще до имплантации эмбриона в матку с точностью до 99,9% диагностировать у него хромосомные аномалии или моногенные заболевания.
Метод NGS используется при экстракорпоральном оплодотворении (ЭКО), значительно повышает вероятность наступления беременности в программе ЭКО, а также существенно снижает вероятность появления на свет малышей с генетическими патологиями.
Суть метода NGS
Метод NGS (Next Generation Sequencing) является частью программы экстракорпорального оплодотворения (ЭКО) и применяется с целью преимплантационной генетической диагностики или скрининга (ПГД или ПГС) эмбрионов. Этот метод представляет собой современную технологию, с помощью которой расшифровывается геном эмбриона. Тест NGS дает очень низкий процент ошибочных результатов.
Зачем нужна преимплантационная генетическая диагностика эмбрионов?
Научные исследования показывают, что матка является очень чутким биосенсором, который распознает эмбрионы, имеющие хромосомные аномалии, и «отказывает» им в имплантации.
До появления метода NGS во многих клиниках, специализирующихся на вспомогательных репродуктивных технологиях, преимплантационная диагностика эмбрионов осуществлялась с помощью других методик, в частности, с помощью метода FISH, который имеет ограниченные возможности диагностики, тестируя лишь небольшое количество хромосом.
Кроме того, так называемые мозаичные эмбрионы, имеющие различный потенциал для имплантации в зависимости от степени мозаичности, не могут быть выявлены при помощи метода FISH. Поэтому клиника ISIDA внедрила у себя высокочувствительный метод NGS.
Этапы NGS-диагностики
Метод NGS является частью программы ЭКО с преимплантационной диагностикой. Обследования и подготовка супругов к экстракорпоральному оплодотворению с применением метода NGS или без его применения ничем не отличаются: врач назначает будущим родителям обследования, основываясь на данных их семейного анамнеза.
Первым этапом ЭКО (не считая предшествующие диагностические исследования) является стимуляция овуляции – введение в организм женщины гормональных препаратов, под влиянием которых в одном менструальном цикле созревает не одна, как обычно, а несколько яйцеклеток. При помощи пункции их извлекают из организма женщины и оплодотворяют спермой будущего папы методом ИКСИ (интрацитоплазматическая инъекция сперматозоида) в лабораторных условиях.
На пятые сутки после оплодотворения клетки эмбриона начинают дифференцироваться, разделяясь на «внутриклеточную массу», из которой сформируется будущий ребенок и на клетки внешнего слоя – трофектодермы, которые примут участие в имплантации эмбриона в полость матки и сформируют плаценту.
Для NGS-анализа берут клетки трофектодермы. Используя высокоточные микроинструменты, врач берет для анализа всего несколько клеток, минимизируя таким образом возможность повреждения эмбриона.
Генетическая диагностика.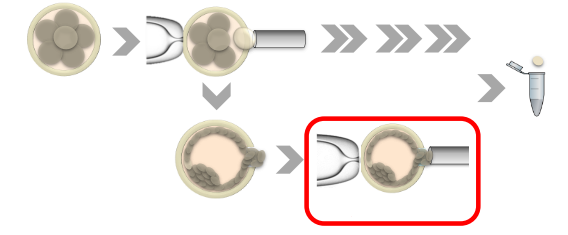 Взятые для анализа клетки передаются в молекулярно-диагностическую лабораторию, в которой с помощью метода NGS проводится анализ эмбриона на наличие хромосомных или генных аномалий. Точность проверки на хромосомные или моногенные заболевания при этом составляет около 99,9%. Процесс диагностики полностью автоматизирован, что является одной из гарантий высокой точности результата.
Взятые для анализа клетки передаются в молекулярно-диагностическую лабораторию, в которой с помощью метода NGS проводится анализ эмбриона на наличие хромосомных или генных аномалий. Точность проверки на хромосомные или моногенные заболевания при этом составляет около 99,9%. Процесс диагностики полностью автоматизирован, что является одной из гарантий высокой точности результата.
Криоцикл для эмбриона. В лабораторных условиях развитие эмбриона ограничено во времени – не более 5-6 дней. Учитывая тот факт, что анализ требует времени, а клетки трофектодермы можно получить только на 5-6 сутки развития эмбриона, после биопсии («отщипывания» небольшого количества клеточек для анализа), эмбрион замораживают современным и эффективным методом витрификации. После получения результатов анализа женщина проходит дополнительную подготовку к криоциклу.
Перенос эмбриона. Перенос эмбрионов, которые после получения результатов анализа NGS признаются нормальными, осуществляется в следующем, запланированном вместе с врачем-репродуктологом, цикле – в периоде, наиболее подходящем для имплантации эмбриона. В ожидании этого времени врачи еще раз проверяют, а при необходимости – стабилизируют гормональный фон будущей мамы, убеждаются в готовности эндометрия к имплантации.
В ожидании этого времени врачи еще раз проверяют, а при необходимости – стабилизируют гормональный фон будущей мамы, убеждаются в готовности эндометрия к имплантации.
После размораживания эмбрионы с помощью абсолютно безболезненной манипуляции под контролем УЗИ переносят в полость матки. Дальнейшее течение беременности и родоразрешение не отличаются от такового после естественного зачатия.
Кому показан метод NGS
Метод NGS рекомендуется в следующих случаях:
Если супруги уже проходили несколько попыток ЭКО и беременность не состоялась, либо имелись множественные замершие беременности, выкидыши на ранних сроках беременности. Большая часть случаев ненаступления беременности связана с наличием у эмбриона хромосомных нарушений. В этом случае возникает довольно высокий риск гибели эмбриона еще до его имплантации и ненаступление в связи с этим беременности. Иногда гибель эмбриона происходит после имплантации – это приводит к выкидышам на ранних сроках беременности или рождению ребенка с хромосомными или генными патологиями.
Проведение преимплантационной диагностики с применением NGS позволяет с высокой вероятностью отобрать генетически здоровый эмбрион и значительно уменьшить таким образом вышеперечисленные риски.
Если супруги знают о наличие в семейном анамнезе генетических заболеваний. В случае, если ближайшие родственники супругов страдают тяжелыми наследственными заболеваниями, выходом может быть метод NGS.
Возраст также является одним из показаний к проведению анализа NGS. Ведь с течением времени вероятность развития хромосомных аномалий, особенно у женщин, значительно увеличивается.
«Одна из самых важных причин ненаступления беременности у женщин в возрасте старше 38 лет – это возрастающая доля наличия хромосомных аномалий в яйцеклетках, – говорит Кременска Юлия Валерьевна, биолог, эмбриолог, молекулярный генетик. – Нужно отметить, что риск появления хромосомных мутаций (анеуплоидий) в яйцеклетке не зависит от способа зачатия. Кроме того, повышенный риск зачатия плода с хромосомными и генетическими мутациями появляется в случае диагностированного носительства генетических заболеваний, наличия в семье генетических заболеваний. Чтобы не допустить аномалий у ребенка и исключить риск необходимости прерывания беременности по медицинским показаниям, ЭКО с преимплантационной диагностикой рекомендовано в таких случаях даже тем парам, которые могут зачать ребенка самостоятельно».
Кроме того, повышенный риск зачатия плода с хромосомными и генетическими мутациями появляется в случае диагностированного носительства генетических заболеваний, наличия в семье генетических заболеваний. Чтобы не допустить аномалий у ребенка и исключить риск необходимости прерывания беременности по медицинским показаниям, ЭКО с преимплантационной диагностикой рекомендовано в таких случаях даже тем парам, которые могут зачать ребенка самостоятельно».
Эффективность ЭКО с NGS
Метод NGS значительно повышает эффективность программы ЭКО. Высокая точность определения хромосомных аномалий (99,9%) позволяет не только избежать риска появления на свет ребенка с тяжелыми наследственными заболеваниями, но и значительно повысить успешность имплантации эмбрионов. Кроме того, метод NGS значительно снижает риск возникновение угрозы выкидыша на ранних сроках беременности. Это состояние, согласно данным доказательной медицины, примерно в половине случаев возникает как следствие наличия генетических аномалий у плода: в действие вступает суровый закон естественного отбора.
К успеху приводит работа команды
Метод NGS является одним из самых ярких проявлений командной работы клиники ISIDA, которая уже 25 лет специализируется на лечении бесплодия и применении методик ВРТ (вспомогательных репродуктивных технологий). Более 10 лет в клинике ISIDA практикуются методы преимплантационной диагностики эмбрионов. Внедрение метода NGS стало возможным благодаря постоянной нацеленности клиники на внедрение в свою практику инновационных методов.
С этой целью репродуктологи, эмбриологи, биологи, генетики клиники регулярно участвуют в работе отечественных и зарубежных научно-практических конференций, семинаров, конгрессов, проходят стажировку в ведущих клиниках мира. Все это дает возможность всегда быть в курсе достижений современной медицины, внедрять у себя последние медицинские инновации, такие как метод NGS.
Преимплантационная генетическая диагностика или скрининг (PGD/PGS) — метод NGS from ISIDA
Для успешной реализации метода NGS необходимо участие гинекологов и репродуктологов, которые определяют необходимость использования этого метода, проводят гормональную стимуляцию овуляции, определяют готовность организма женщины к имплантации, проводят манипуляцию подсадки эмбрионов.
Особая ответственность возлагается на биологов и эмбриологов, которые в лабораторных условиях осуществляют оплодотворение яйцеклетки, культивирование эмбрионов, проводят процедуру биопсии эмбриона, а также реализуют метод витрификации. Работа с гаметами и эмбрионами требует не только высокого профессионализма, но и чуткого к чужим мечтам и надеждам сердца. 25 лет работы и более 20 000 появившихся на свет, несмотря на самые сложные диагнозы малышей – таковы вехи слаженной командной работы специалистов клиники ISIDA.
Хотите, чтобы Ваши мечты о материнстве стали счастливой реальностью? Доверьте свое счастье профессионалам: позвоните по телефонам 0 800 60 80 80, +38 (044) 455 88 11 и запишитесь на прием к врачу, который поддержит Вас на пути к Вашей мечте. Счастливого Вам материнства!
Технология секвенирования следующего поколения в клинике и ее проблемы
1. Мецкер М.Л. Технологии секвенирования — следующее поколение. Нац. Преподобный Жене. 2010;11:31–46. doi: 10.1038/nrg2626. [PubMed] [CrossRef] [Google Scholar]
2010;11:31–46. doi: 10.1038/nrg2626. [PubMed] [CrossRef] [Google Scholar]
2. Лангмид Б., Зальцберг С.Л. Быстрое выравнивание с промежутками чтения с Bowtie 2. Nat. Методы. 2012; 9: 357–359. doi: 10.1038/nmeth.1923. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
3. Ли Х., Дурбин Р. Быстрое и точное выравнивание коротких прочтений с помощью преобразования Берроуза-Уилера. Биоинформатика. 2009 г.;25:1754–1760. doi: 10.1093/биоинформатика/btp324. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
4. Бенджамин Д., Сато Т., Цибульскис К., Гетц Г., Стюарт К., Лихтенштейн Л. Вызов соматических SNV и Indels с помощью Mutect2. bioRxiv. 2019:861054. дои: 10.1101/861054. [CrossRef] [Google Scholar]
5. Ким С., Шеффлер К., Халперн А.Л., Бекрицкий М.А., Но Э., Келлберг М., Чен Х., Ким Ю., Бейтер Д., Круше П. и др. др. Стрелка2: Быстрый и точный вызов зародышевых и соматических вариантов. Нац. Методы. 2018;15:591–594. doi: 10.1038/s41592-018-0051-x. [PubMed] [CrossRef] [Google Scholar]
[PubMed] [CrossRef] [Google Scholar]
6. Kumaran M., Subramanian U., Devarajan B. Оценка производительности конвейеров вызова вариантов с использованием секвенирования всего экзома человека и смоделированных данных. БМК Биоинформ. 2019;20:342. doi: 10.1186/s12859-019-2928-9. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
7. Liu X., Han S., Wang Z., Gelernter J., Yang B.Z. Вызывающие варианты для данных секвенирования следующего поколения: сравнительное исследование. ПЛОС ОДИН. 2013;8:e75619. doi: 10.1371/journal.pone.0075619. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
8. Максам А.М., Гилберт В. Новый метод секвенирования ДНК. проц. Натл. акад. науч. США. 1977; 74: 560–564. doi: 10.1073/pnas.74.2.560. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
9. Sanger F., Nicklen S., Coulson A.R. Секвенирование ДНК с помощью ингибиторов обрыва цепи. проц. Натл. акад. науч. США. 1977; 74: 5463–5467. doi: 10.1073/pnas.74. 12.5463. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
12.5463. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10. Олсон М.В. Проект генома человека. проц. Натл. акад. науч. США. 1993; 90:4338–4344. doi: 10.1073/pnas.90.10.4338. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
11. Хизер Дж. М., Цепь Б. Последовательность секвенаторов: история секвенирования ДНК. Геномика. 2016; 107:1–8. doi: 10.1016/j.ygeno.2015.11.003. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
12. Слатко Б.Е., Гарднер А.Ф., Аусубель Ф.М. Обзор технологий секвенирования следующего поколения. Курс. протокол Мол. биол. 2018;122:e59. doi: 10.1002/cpmb.59. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
13. Сяо Т., Чжоу В. Секвенирование третьего поколения: передовой подход к генетическим заболеваниям. Перевод Педиатр. 2020; 9: 163–173. doi: 10.21037/tp.2020.03.06. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
14. Pertea M., Shumate A., Pertea G., Varabyou A., Breitwieser F. P., Chang Y., Madugundu A.K., Pandey A., Salzberg Л.С. ШАХМАТЫ: Новый каталог генов человека, созданный на основе тысяч крупномасштабных экспериментов по секвенированию РНК, обнаруживает обширный транскрипционный шум. Геном биол. 2018;19:208. doi: 10.1186/s13059-018-1590-2. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
P., Chang Y., Madugundu A.K., Pandey A., Salzberg Л.С. ШАХМАТЫ: Новый каталог генов человека, созданный на основе тысяч крупномасштабных экспериментов по секвенированию РНК, обнаруживает обширный транскрипционный шум. Геном биол. 2018;19:208. doi: 10.1186/s13059-018-1590-2. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
15. Pon J.R., Marra M.A. Мутации водителя и пассажира при раке. Анну. Преподобный Патол. мех. Дис. 2015;10:25–50. doi: 10.1146/annurev-pathol-012414-040312. [PubMed] [CrossRef] [Google Scholar]
16. МакФарланд С.Д., Мирный Л.А., Королев К.С. Перетягивание каната между мутациями водителя и пассажира при раке и других адаптивных процессах. проц. Натл. акад. науч. США. 2014;111:15138–15143. doi: 10.1073/pnas.1404341111. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
17. Futreal P.A., Coin L., Marshall M., Down T., Hubbard T., Wooster R., Rahman N., Stratton M.R. Перепись генов рака человека. Нац. Преподобный Рак. 2004; 4: 177–183. doi: 10.1038/nrc1299. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
doi: 10.1038/nrc1299. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
18. Дитлайн Ф., Вегхорн Д., Тейлор-Вайнер А., Рихтерс А., Рирдон Б., Лю Д., Ландер Э.С., Ван Аллен Е.М., Сюняев С.Р. Идентификация генов-драйверов рака на основе нуклеотидного контекста. Нац. Жене. 2020; 52: 208–218. doi: 10.1038/s41588-019-0572-й. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
19. Bailey M.H., Tokheim C., Porta-Pardo E., Sengupta S., Bertrand D., Weerasinghe A., Colaprico A., Wendl M.C. , Ким Дж., Рирдон Б. и др. Всесторонняя характеристика генов и мутаций, вызывающих рак. Клетка. 2018;173:371–385. doi: 10.1016/j.cell.2018.02.060. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
20. Гонсалес-Перес А., Мустонен В., Рева Б., Ричи Г.Р.С., Крейкселл П., Карчин Р., Васкес М., Финк Дж.Л. , Kassahn K.S., Pearson J.V., et al. Вычислительные подходы к идентификации функциональных генетических вариантов в геномах рака. Нац. Методы. 2013;10:723–729. doi: 10.1038/nmeth.2562. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
doi: 10.1038/nmeth.2562. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
21. Кумар К.Р., Коули М.Дж., Дэвис Р.Л. Секвенирование нового поколения и новые технологии. Семин. тромб. Хемост. 2019;45:661–673. doi: 10.1055/s-0039-1688446. [PubMed] [CrossRef] [Google Scholar]
22. Шин С.Х., Боде А.М., Донг З. Точная медицина: основа будущей терапии рака. Npj Precis. Онкол. 2017;1:12. doi: 10.1038/s41698-017-0016-z. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
23. Кортес А.Дж., Тудрей П., Куява К.А., Лисовска К.М. Достижения в терапии рака яичников. Рак Чемотер. Фармакол. 2018;81:17–38. doi: 10.1007/s00280-017-3501-8. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
24. Caulfield S.E., Davis C.C., Byers K.F. Олапариб: новая терапия метастатического рака молочной железы у пациентов с мутацией BRCA1/2. Дж. Адв. Практика. Онкол. 2019;10:167–174. doi: 10.6004/jadpro.2019.10.2.6. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
25. DeMatteo R.P., Ballman K.V., Antonescu C.R., Make R.G., Pisters P.W.T., Demetri G.D., Blackstein M.E., Blanke C.D., Von Mehren M., Brennan M.F., et al. Адъювантный мезилат иматиниба после резекции локализованной первичной стромальной опухоли желудочно-кишечного тракта: рандомизированное двойное слепое плацебо-контролируемое исследование. Ланцет. 2009; 373:1097–1104. doi: 10.1016/S0140-6736(09)60500-6. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
DeMatteo R.P., Ballman K.V., Antonescu C.R., Make R.G., Pisters P.W.T., Demetri G.D., Blackstein M.E., Blanke C.D., Von Mehren M., Brennan M.F., et al. Адъювантный мезилат иматиниба после резекции локализованной первичной стромальной опухоли желудочно-кишечного тракта: рандомизированное двойное слепое плацебо-контролируемое исследование. Ланцет. 2009; 373:1097–1104. doi: 10.1016/S0140-6736(09)60500-6. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
26. Чепмен П.Б., Хаушильд А., Роберт С., Хаанен Дж.Б., Асьерто П., Ларкин Дж., Даммер Р., Гарбе С., Тестори А., Майо М. и др. Улучшение выживаемости при применении вемурафениба при меланоме с мутацией BRAF V600E. Н. англ. Дж. Мед. 2011; 364:2507–2516. doi: 10.1056/NEJMoa1103782. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
27. Дину Д., Добре М., Панайтеску Э., Бирла Р., Иосиф К., Хоара П., Карагуи А., Боэриу М., Константиною С., Арделяну К. Прогностическое значение мутаций гена KRAS у колоректальный рак — предварительное исследование. Дж. Мед. Жизнь. 2014;7:581–587. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дж. Мед. Жизнь. 2014;7:581–587. [Бесплатная статья PMC] [PubMed] [Google Scholar]
28. Maus M.K.H., Grimminger PP, Mack PC, Astrow S.H., Stephens C., Zeger G., Hsiang J., Brabender J., Friedrich M., Alakus H. ., и другие. Мутации KRAS при немелкоклеточном раке легкого и колоректальном раке: значение для терапии, нацеленной на EGFR. Рак легких. 2014; 83: 163–167. doi: 10.1016/j.lungcan.2013.11.010. [PubMed] [CrossRef] [Академия Google]
29. Forbes S.A., Beare D., Boutselakis H., Bamford S., Bindal N., Tate J., Cole C.G., Ward S., Dawson E., Ponting L., et al. COSMIC: Генетика соматического рака в высоком разрешении. Нуклеиновые Кислоты Res. 2017; 45:777–783. doi: 10.1093/nar/gkw1121. [Статья PMC бесплатно] [PubMed] [CrossRef] [Google Scholar]
30. Tate J.G., Bamford S., Jubb H.C., Sondka Z., Beare DM, Bindal N., Boutselakis H., Cole C.G., Creatore C. , Доусон Э. и др. КОСМИЧЕСКИЙ: Каталог соматических мутаций при раке. Нуклеиновые Кислоты Res. 2019;47:941–947. doi: 10. 1093/nar/gky1015. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
1093/nar/gky1015. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
ClinVar: общедоступный архив взаимосвязей между вариациями последовательности и фенотипом человека. Нуклеиновые Кислоты Res. 2014;42:980–985. doi: 10.1093/nar/gkt1113. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
32. Ландрам М.Дж., Ли Дж.М., Бенсон М., Браун Г.Р., Чао С., Читипиралла С., Гу Б., Харт Дж., Хоффман Д. ., Джанг В. и др. ClinVar: Улучшение доступа к различным интерпретациям и подтверждающим доказательствам. Нуклеиновые Кислоты Res. 2018;46:1062–1067. дои: 10.1093/нар/gkx1153. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
33. Chakravarty D., Gao J., Phillips S., Kundra R., Zhang H., Wang J., Rudolph J.E., Yaeger R., Сумерай Т., Ниссан М.Х. и др. OncoKB: База знаний по прецизионной онкологии. JCO Precis. Онкол. 2017; 1:1–16. doi: 10.1200/PO.17.00011. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
34. Bewicke-Copley F., Arjun Kumar E. , Palladino G., Korfi K., Wang J. Применение и анализ целевого геномного секвенирования при раке исследования. вычисл. Структура Биотехнолог. Дж. 2019;17:1348–1359. doi: 10.1016/j.csbj.2019.10.004. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
, Palladino G., Korfi K., Wang J. Применение и анализ целевого геномного секвенирования при раке исследования. вычисл. Структура Биотехнолог. Дж. 2019;17:1348–1359. doi: 10.1016/j.csbj.2019.10.004. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
35. Гриффит М., Миллер К.А., Гриффит О.Л., Крысяк К., Скидмор З.Л., Раму А., Уокер Дж.Р., Данг Х.Х., Трани Л., Ларсон Д.Э. и соавт. Оптимизация секвенирования и анализа генома рака. Сотовая система 2015;1:210–223. doi: 10.1016/j.cels.2015.08.015. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
36. Williams C., Pontén F., Moberg C., Söderkvist P., Uhlén M., Pontén J., Sitbon G., Lundeberg J. Высокая частота изменений последовательностей обусловлена формалиновой фиксацией архивных препаратов. Являюсь. Дж. Патол. 1999;155:1467–1471. doi: 10.1016/S0002-9440(10)65461-2. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
37. Kim S., Park C., Ji Y., Kim D.G., Bae H., Vrancken M., Kim D., Kim K. Дезаминирование Эффекты фиксированных формалином и залитых парафином образцов тканей в эпоху прецизионной медицины. Дж. Мол. Диагн. 2017;19:137–146. doi: 10.1016/j.jmoldx.2016.09.006. [PubMed] [CrossRef] [Google Scholar]
Дезаминирование Эффекты фиксированных формалином и залитых парафином образцов тканей в эпоху прецизионной медицины. Дж. Мол. Диагн. 2017;19:137–146. doi: 10.1016/j.jmoldx.2016.09.006. [PubMed] [CrossRef] [Google Scholar]
38. Gao X.H., Li J., Gong H.F., Yu G.Y., Liu P., Hao L.Q., Liu L.J., Bai C.G., Zhang W. Сравнение свежезамороженной ткани с формалином — Фиксированная ткань, залитая парафином, для анализа мутаций с использованием мультигенной панели у пациентов с колоректальным раком. Фронт. Онкол. 2020; 10:1–8. дои: 10.3389/fonc.2020.00310. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
39. Керик М., Исау М., Тиммерманн Б., Зультманн Х., Хервиг Р., Кробич С., Шефер Г., Вердорфер И. , Bartsch G., Klocker H., et al. Направленное высокопроизводительное секвенирование при клиническом раке. Параметры: опухолевые ткани, залитые формальдегидом в фиксированный парафин (FFPE), количество вводимых данных и гетерогенность опухоли. БМС Мед. Геном. 2011; 4:1–13. дои: 10. 1186/1755-8794-4-68. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
1186/1755-8794-4-68. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
40. Баст Р.К., Фини М., Лазарус Х., Надлер Л.М., Колвин Р.Б., Кнапп Р.К. Реактивность моноклонального антитела с карциномой яичников человека. Дж. Клин. расследование 1981; 68: 1331–1337. doi: 10.1172/JCI110380. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
41. Локтионов А. Биомаркеры для неинвазивного выявления колоректального рака: ДНК, РНК или белки. Мир J. Гастроинтест. Онкол. 2020;12:124–128. doi: 10.4251/wjgo.v12.i2.124. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
42. Мамдани Х., Ахмед С., Армстронг С., Мок Т., Джалал С.И. Опухолевые биомаркеры на основе крови при раке легкого для обнаружения и лечения. Перевод Рак легких Res. 2017; 6: 648–660. doi: 10.21037/tlcr.2017.09.03. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
43. Олооми М., Моаззези Н., Бузари С. Сравнение экспрессии биомаркеров крови и тканей у пациентов с раком молочной железы. Гелион. 2020;6:1–7. doi: 10.1016/j.heliyon.2020.e03728. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Гелион. 2020;6:1–7. doi: 10.1016/j.heliyon.2020.e03728. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
44. Дагого-Джек И., Шоу А.Т. Гетерогенность опухоли и резистентность к терапии рака. Нац. Преподобный Клин. Онкол. 2018;15:81–94. doi: 10.1038/nrclinonc.2017.166. [PubMed] [CrossRef] [Google Scholar]
45. Келлер Л., Беллум Ю., Викман Х., Пантел К. Клиническая значимость анализа цДНК крови: обнаружение мутаций и не только. бр. Дж. Рак. 2020; 6:1–14. doi: 10.1038/s41416-020-01047-5. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
46. Mouliere F., Chandrananda D., Piskorz A.M., Moore E.K., Morris J., Ahlborn L.B., Mair R., Goranova T., Marass F ., Хайдер К. и др. Расширенное обнаружение циркулирующей опухолевой ДНК с помощью анализа размера фрагмента. науч. Перевод Мед. 2018; 10:1–14. doi: 10.1126/scitranslmed.aat4921. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
47. Ignatiadis M., Dawson S.J. Циркулирующие опухолевые клетки и циркулирующая опухолевая ДНК для прецизионной медицины: мечта или реальность? Анна. Онкол. 2014;25:2304–2313. doi: 10.1093/annonc/mdu480. [PubMed] [CrossRef] [Google Scholar]
Онкол. 2014;25:2304–2313. doi: 10.1093/annonc/mdu480. [PubMed] [CrossRef] [Google Scholar]
48. Дженнингс Л.Дж., Арсила М.Е., Корлесс С., Камел-Рейд С., Лубин И.М., Пфайфер Дж., Темпл-Смолкин Р.Л., Фелькердинг К.В., Никифорова М.Н. Руководство по валидации онкологических панелей на основе секвенирования следующего поколения: совместная согласованная рекомендация Ассоциации молекулярной патологии и Колледжа американских патологов. Дж. Мол. Диагн. 2017;19: 341–365. doi: 10.1016/j.jmoldx.2017.01.011. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
49. Xu C. Обзор алгоритмов вызова соматических однонуклеотидных вариантов для данных секвенирования следующего поколения. вычисл. Структура Биотехнолог. Ж. 2018; 16:15–24. doi: 10.1016/j.csbj.2018.01.003. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
50. Treangen T.J., Salzberg S.L. Повторяющаяся ДНК и секвенирование следующего поколения: вычислительные проблемы и решения. Нац. Преподобный Жене. 2012; 13:36–46. doi: 10.1038/nrg3117. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
2012; 13:36–46. doi: 10.1038/nrg3117. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
51. Lee C.Y., Yen H.Y., Zhong A.W., Gao H. Устранение помех смещения для клинической диагностики на основе NGS. Гум. Жене. 2020; 9:1–16. doi: 10.1007/s00439-020-02216-5. [PubMed] [CrossRef] [Google Scholar]
52. Pink R.C., Wicks K., Caley D.P., Punch E.K., Jacobs L., Carter D.R.F. Псевдогены: псевдофункциональные или ключевые регуляторы здоровья и болезней? РНК. 2011; 11: 792–798. doi: 10.1261/РНК.2658311. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
53. Хань Л., Юань Ю., Чжэн С., Ян Ю., Ли Дж., Эдгертон М.Е., Дяо Л., Сюй Ю., Verhaak R.G.W., Liang H. Панраковый анализ экспрессии псевдогена выявляет биологически и клинически значимые подтипы опухолей. Нац. коммун. 2014; 5:1–9. doi: 10.1038/ncomms4963. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
54. Puget N., Gad S., Perrin-Vidoz L., Sinilnikova O.M., Stoppa-Lyonnet D., Lenoir G. M., Mazoyer S. Distinct BRCA1 перестройки с участием псевдогена BRCA1 предполагают существование горячей точки рекомбинации. Являюсь. Дж. Хам. Жене. 2002; 70: 858–865. дои: 10.1086/339434. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
M., Mazoyer S. Distinct BRCA1 перестройки с участием псевдогена BRCA1 предполагают существование горячей точки рекомбинации. Являюсь. Дж. Хам. Жене. 2002; 70: 858–865. дои: 10.1086/339434. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
55. Перейра Р., Оливейра Дж., Соуза М. Биоинформатика и вычислительные инструменты для анализа секвенирования следующего поколения в клинической генетике. Дж. Клин. Мед. 2020;9:132. doi: 10.3390/jcm
32. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]56. Meynert A.M., Ansari M., FitzPatrick D.R., Taylor M.S. Чувствительность обнаружения вариантов и погрешности при секвенировании всего генома и экзома. БМК Биоинформ. 2014; 15:1–11. дои: 10.1186/1471-2105-15-247. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
57. Корниш А., Гуда С. Сравнение конвейеров вызова вариантов с использованием генома в бутылке в качестве эталона. Биомед Рез. Междунар. 2015;456479: 1–11. doi: 10.1155/2015/456479. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
58. Guo Y., Dai Y., Yu H., Zhao S., Samuels D.C., Shyr Y. Улучшения и влияние человеческого эталона GRCh48 на высокие анализ данных секвенирования пропускной способности. Геномика. 2017;109:83–90. doi: 10.1016/j.ygeno.2017.01.005. [PubMed] [CrossRef] [Google Scholar]
59. Pan B., Kusko R., Xiao W., Zheng Y., Liu Z., Xiao C., Sakkiah S., Guo W., Gong P., Чжан С. и др. Сходства и различия между вариантами, названными эталонным геномом человека HG19.или HG38. БМК Биоинформ. 2019;20:17–29. doi: 10.1186/s12859-019-2620-0. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
60. Yu X., Guda K., Willis J., Veigl M., Wang Z., Markowitz S., Adams MD, Sun S. How работают ли программы выравнивания с данными секвенирования различного качества и из повторяющихся областей? Биоданные Мин. 2012; 5:1–12. дои: 10.1186/1756-0381-5-6. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
61. Chen J. , Li X., Zhong H., Meng Y., Du H. Систематическое сравнение вариантов зародышевой линии, вызывающих конвейеры, пересекающих несколько каналов следующего поколения секвенсоры. науч. Респ. 2019 г.;9:1–13. doi: 10.1038/s41598-019-45835-3. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
, Li X., Zhong H., Meng Y., Du H. Систематическое сравнение вариантов зародышевой линии, вызывающих конвейеры, пересекающих несколько каналов следующего поколения секвенсоры. науч. Респ. 2019 г.;9:1–13. doi: 10.1038/s41598-019-45835-3. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
62. Chen Z., Yuan Y., Chen X., Chen J., Lin S., Li X., Du H. Систематическое сравнение соматических вариант вызова производительности среди различной глубины секвенирования и частоты мутаций. науч. Отчет 2020; 10: 1–9. doi: 10.1038/s41598-020-60559-5. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Сравнительный анализ конвейеров полногеномного секвенирования для минимизации ложноотрицательных результатов. науч. Респ. 2019 г.;9:1–10. doi: 10.1038/s41598-019-39108-2. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
64. Хван С., Ким Э., Ли И., Маркотт Э. М. Систематическое сравнение конвейеров вызовов вариантов с использованием вариантов персонального экзома золотого стандарта. науч. Отчет 2015; 5: 1–8. doi: 10.1038/srep17875. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
науч. Отчет 2015; 5: 1–8. doi: 10.1038/srep17875. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
65. Meng J., Chen Y.P.P. База данных смоделированных геномов опухолей для точного обнаружения малых соматических вариантов рака. ПЛОС ОДИН. 2018;13:e202982. doi: 10.1371/journal.pone.0202982. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
66. Супернат А., Видарссон О.В., Стин В.М., Стокови Т. Сравнение трех вариантов вызывающих для секвенирования всего генома человека. науч. Отчет 2018; 8: 1–6. doi: 10.1038/s41598-018-36177-7. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
67. Xu H., DiCarlo J., Satya R.V., Peng Q., Wang Y. Сравнение методов вызова соматических мутаций в данных последовательности ампликона и полного экзома . БМС Геном. 2014; 15:1–10. дои: 10.1186/1471-2164-15-244. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
68. Mielczarek M., Szyda J. Обзор алгоритмов выравнивания и вызова SNP для данных секвенирования следующего поколения. Дж. Заявл. Жене. 2016;57:71–79. doi: 10.1007/s13353-015-0292-7. [PubMed] [CrossRef] [Google Scholar]
Дж. Заявл. Жене. 2016;57:71–79. doi: 10.1007/s13353-015-0292-7. [PubMed] [CrossRef] [Google Scholar]
69. Kuhnle A., Mun T., Boucher C., Gagie T., Langmead B., Manzini G. Эффективное построение полного индекса для пангеномного выравнивания чтения. Дж. Вычисл. биол. 2020;27:500–513. doi: 10.1089/cmb.2019.0309. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
70. Линднер Р., Фридель К.К. Комплексная оценка алгоритмов выравнивания в контексте RNA-Seq. ПЛОС ОДИН. 2012;7:e52403. doi: 10.1371/journal.pone.0052403. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
71. Zhang H., Chan Y., Fan K., Schmidt B., Liu W. Быстрое и эффективное сопоставление краткого чтения на основе краткого хэш-индекса . БМК Биоинформ. 2018;19:1–14. doi: 10.1186/s12859-018-2094-5. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
72. Liu Y., Popp B., Schmidt B. CUSHAW3: чувствительное и точное выравнивание коротких считываний в базовом и цветовом пространстве с гибридным заполнением. ПЛОС ОДИН. 2014;9:e86869. doi: 10.1371/journal.pone.0086869. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
ПЛОС ОДИН. 2014;9:e86869. doi: 10.1371/journal.pone.0086869. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
73. Wu T.D., Nuca S. Быстрое и устойчивое к SNP обнаружение сложных вариантов и сплайсинг в коротких чтениях. Биоинформатика. 2010; 26: 873–881. doi: 10.1093/биоинформатика/btq057. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
74. Raczy C., Petrovski R., Saunders C.T., Chorny I., Kruglyak S., Margulies E.H., Chuang H., Källberg M., Kumar С.А., Ляо А. и др. Исаак: Сверхбыстрый вторичный анализ всего генома на платформах секвенирования Illumina. Биоинформатика. 2013;29: 2041–2043. doi: 10.1093/биоинформатика/btt314. [PubMed] [CrossRef] [Google Scholar]
75. Lee W.P., Stromberg M.P., Ward A., Stewart C., Garrison E.P., Marth G.T. MOSAIK: Алгоритм на основе хэшей для точного картирования коротких прочтений секвенирования следующего поколения. ПЛОС ОДИН. 2014;9:e
. doi: 10.1371/journal.pone.00. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar] 76. Li R., Yu C., Li Y., Lam T., Yiu S., Kristiansen K., Wang J. SOAP2: улучшенный сверхбыстрый инструмент для выравнивания коротких прочтений. Биоинформатика. 2009 г.;25:1966–1967. doi: 10.1093/биоинформатика/btp336. [PubMed] [CrossRef] [Google Scholar]
Li R., Yu C., Li Y., Lam T., Yiu S., Kristiansen K., Wang J. SOAP2: улучшенный сверхбыстрый инструмент для выравнивания коротких прочтений. Биоинформатика. 2009 г.;25:1966–1967. doi: 10.1093/биоинформатика/btp336. [PubMed] [CrossRef] [Google Scholar]
77. Challis D., Yu J., Evani U.S., Jackson A.R., Paithankar S., Coarfa C., Milosavljevic A., Gibbs R.A., Yu F. Интегративный анализ вариантов набор для данных секвенирования нового поколения всего экзома. БМК Биоинформ. 2012; 8:1–12. дои: 10.1186/1471-2105-13-8. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
ПРОТИВ: анализ числа копий для целевого изменения последовательности. Биоинформатика. 2012; 28:1307–1313. дои: 10.1093/биоинформатика/bts146. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
79. Абызов А., Урбан А.Е., Снайдер М., Герштейн М. CNVnator: Подход к обнаружению, генотипированию и характеристике типичных и атипичных CNV из семьи и секвенирование популяционного генома. Геном Res. 2011;21:974–984. doi: 10.1101/gr.114876.110. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
2011;21:974–984. doi: 10.1101/gr.114876.110. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
80. Amarasinghe KC, Li J., Halgamuge S.K. Исправление к CoNVEX: оценка вариации числа копий в данных секвенирования экзома с использованием HMM. БМК Биоинформ. 2013; 14:1–9. doi: 10.1186/1471-2105-14-S2-S26. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
81. Poplin R., Chang P., Alexander D., Schwartz S., Colthurst T., Ku A., Newburger D., Dijamco J. , Нгуен Н., Афшар П.Т. и др. Универсальный вызывающий модуль snp и small-indel, использующий глубокие нейронные сети. Нац. Биотехнолог. 2018; 36: 983–987. doi: 10.1038/nbt.4235. [PubMed] [CrossRef] [Google Scholar]
82. Rausch T., Zichner T., Schlattl A., Stütz A.M., Benes V., Korbel J.O. DELLY: обнаружение структурных вариантов с помощью интегрированного анализа парных концов и раздельного чтения. Биоинформатика. 2012; 28: 333–339.. doi: 10.1093/биоинформатика/bts378. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Изменение количества копий на основе секвенирования экзома и обнаружение потери гетерозиготности: ExomeCNV. Биоинформатика. 2011;27:2648–2654. doi: 10.1093/биоинформатика/btr462. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Биоинформатика. 2011;27:2648–2654. doi: 10.1093/биоинформатика/btr462. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
84. Гаррисон Э., Март Г. Обнаружение вариантов на основе гаплотипов с помощью короткого секвенирования. архив 20121207,3907 [Google Scholar]
85. Маккенна А., Ханна М., Бэнкс Э., Сиваченко А., Цибульскис К., Керницкий А., Гаримелла К., Альтшулер Д., Габриэль С., Дали М. и др. др. Набор инструментов для анализа генома: платформа MapReduce для анализа данных секвенирования ДНК нового поколения. Геном Res. 2009;20:1297–1303. doi: 10.1101/gr.107524.110. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
86. Рен С., Бертельс К., Аль Арс З. Эффективное ускорение алгоритма пересылки пар-HMM для GATK HaplotypeCaller на графических процессорах. Эвол. Биоинформ. 2018; 14:1–12. дои: 10.1177/1176934318760543. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
87. Layer RM, Chiang C., Quinlan AR, Hall IM LUMPY: вероятностная структура для обнаружения структурных вариантов. Геном биол. 2014; 15:1–19. doi: 10.1186/gb-2014-15-6-r84. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
Геном биол. 2014; 15:1–19. doi: 10.1186/gb-2014-15-6-r84. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
сборка. Биоинформатика. 2012;28:3195–3202. doi: 10.1093/биоинформатика/bts601. [PubMed] [CrossRef] [Google Scholar]
89. Цибульскис К., Лоуренс М.С., Картер С.Л., Сиваченко А., Джаффе Д., Сугнез С., Габриэль С., Мейерсон М., Ландер Э.С., Гетц Г. Чувствительное обнаружение соматических точечных мутаций в неочищенных и гетерогенных образцах рака. Нац. Биотехнолог. 2013;31:213–219. doi: 10.1038/nbt.2514. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
90. Ye K., Schulz M.H., Long Q., Apweiler R., Ning Z. Pindel: подход к росту паттерна для обнаружения точек разрыва больших делеций и вставки среднего размера из коротких чтений с парными концами. Биоинформатика. 2009 г.;25:2865–2871. doi: 10.1093/биоинформатика/btp394. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
91. Rimmer A., Phan H., Mathieson I., Iqbal Z., Twigg S. R.F., Wilkie A.O.M., McVean G., Lunter G. Integrating mapping — подходы на основе сборки и гаплотипа для вызова вариантов в приложениях клинического секвенирования. Нац. Жене. 2014;46:912–918. doi: 10.1038/ng.3036. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
R.F., Wilkie A.O.M., McVean G., Lunter G. Integrating mapping — подходы на основе сборки и гаплотипа для вызова вариантов в приложениях клинического секвенирования. Нац. Жене. 2014;46:912–918. doi: 10.1038/ng.3036. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
92. Li H., Handsaker B., Wysoker A., Fennell T., Ruan J., Homer N., Marth G., Abecasis G. , Дурбин Р. Формат Sequence Alignment/Map и SAMtools. Биоинформатика. 2009 г.;25:2078–2079. doi: 10.1093/биоинформатика/btp352. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
93. O’Fallon B.D., Wooderchak-Donahue W., Crockett D.K. Машина опорных векторов для идентификации однонуклеотидных полиморфизмов по данным секвенирования следующего поколения. Биоинформатика. 2013;29:1361–1366. doi: 10.1093/биоинформатика/btt172. [PubMed] [CrossRef] [Google Scholar]
94. Ларсон Д.Э., Харрис К.С., Чен К., Кобольдт Д.К., Эбботт Т.Е., Дулинг Д.Дж., Лей Т.Дж., Мардис Э.Р., Уилсон Р.К., Дин Л. Somaticsniper: Identification of somatic точечные мутации в данных полногеномного секвенирования. Биоинформатика. 2012; 28:311–317. дои: 10.1093/биоинформатика/бтр665. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Биоинформатика. 2012; 28:311–317. дои: 10.1093/биоинформатика/бтр665. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
95. Chiang C., Layer R.M., Faust G.G., Lindberg M.R., Rose DB, Garrison EP, Marth G.T., Quinlan AR, Hall IM SpeedSeq: Ultra- быстрый персональный анализ генома и интерпретация. Нац. Методы. 2015;12:966–968. doi: 10.1038/nmeth.3505. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
96. Saunders C.T., Wong W.S.W., Swamy S., Becq J., Murray L.J., Cheetham R.K. Стрелка: Точный вызов соматических малых вариантов из секвенированных пар опухолевых и нормальных образцов. Биоинформатика. 2012; 28:1811–1817. дои: 10.1093/биоинформатика/bts271. [PubMed] [CrossRef] [Google Scholar]
97. Вонг К., Кин Т.М., Сталкер Дж., Адамс Д.Дж. Усовершенствованный структурный вариант и обнаружение точек останова с помощью SVMerge за счет интеграции нескольких методов обнаружения и локальной сборки. Геном биол. 2010; 11:1–9. doi: 10.1186/gb-2010-11-12-r128. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
98. Gillet-Markowska A., Richard H., Fischer G., Lafontaine I. Ulysses: точное обнаружение низкочастотных структурных вариаций в больших вставках. размер библиотек секвенирования. Биоинформатика. 2015; 31:801–808. дои: 10.1093/биоинформатика/btu730. [PubMed] [CrossRef] [Google Scholar]
99. Koboldt D.C., Zhang Q., Larson D.E., Shen D., McLellan MD, Lin L., Miller C.A., Mardis E.R., Ding L., Wilson R.K. VarScan 2: обнаружение соматических мутаций и изменений количества копий при раке с помощью секвенирования экзома. Геном Res. 2012; 22: 568–576. doi: 10.1101/gr.129684.111. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
100. Бероухим Р., Мермель С.Х., Портер Д., Вей Г., Райчаудхури С., Донован Дж., Барретина Дж., Бём Дж.С., Добсон Дж., Урасима М. и др. Ландшафт соматического изменения числа копий при раке человека. Природа. 2010;463:899–905. doi: 10.1038/nature08822. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
101. Li Y., Roberts N.D., Wala J.A., Shapira O., Schumacher S.E., Kumar K., Khurana E., Waszak S., Korbel J.O. , Haber J.E., et al. Закономерности соматической структурной изменчивости в геномах рака человека. Природа. 2020; 578: 112–121. doi: 10.1038/s41586-019-1913-9. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Li Y., Roberts N.D., Wala J.A., Shapira O., Schumacher S.E., Kumar K., Khurana E., Waszak S., Korbel J.O. , Haber J.E., et al. Закономерности соматической структурной изменчивости в геномах рака человека. Природа. 2020; 578: 112–121. doi: 10.1038/s41586-019-1913-9. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
102. Маротта М., Чен Х., Иношита А., Стивенс Р., Бадд Г.Т., Кроу Дж.П., Лайонс Дж., Кондратова А., Таббс Р., Танака Х. Обычная контрольная точка количества копий амплификации ERBB2 при раке молочной железы колокализируется со сложным блоком сегментарных дупликаций. Рак молочной железы Res. 2012; 14:1–19. doi: 10.1186/bcr3362. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
103. Демиденко З.Н., Фойо Т., Благосклонный М.В. Комплементация двух мутантов p53: последствия потери гетерозиготности при раке. ФЭБС лат. 2005; 579: 2231–2235. doi: 10.1016/j.febslet.2005.03.012. [PubMed] [CrossRef] [Google Scholar]
104. Алаеи-Махабади Б., Бхадури Дж. , Карлссон Дж.В., Нильссон Дж.А., Ларссон Э. Глобальный анализ соматических структурных геномных изменений и их влияние на экспрессию генов при различных видах рака человека. . проц. Натл. акад. науч. США. 2016;113:13768–13773. doi: 10.1073/pnas.1606220113. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
, Карлссон Дж.В., Нильссон Дж.А., Ларссон Э. Глобальный анализ соматических структурных геномных изменений и их влияние на экспрессию генов при различных видах рака человека. . проц. Натл. акад. науч. США. 2016;113:13768–13773. doi: 10.1073/pnas.1606220113. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
105. Алкан С., Коу Б.П., Эйхлер Э.Е. Открытие структурных вариаций генома и генотипирование. Нац. Преподобный Жене. 2011;12:363–376. doi: 10.1038/nrg2958. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
106. Koboldt D.C. Передовой опыт определения вариантов при клиническом секвенировании. Геном Мед. 2020; 12:1–13. doi: 10.1186/s13073-020-00791-w. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
107. Le Scouarnec S., Gribble S.M. Характеристика хромосомных перестроек: последние технические достижения в области молекулярной цитогенетики. Наследственность. 2012; 108:75–85. doi: 10.1038/hdy.2011.100. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
108. Escaramís G., Docampo E., Rabionet R. Десятилетие структурных вариантов: описание, история и методы обнаружения структурных изменений. Краткий. Функц. Геном. 2015;14:305–314. doi: 10.1093/bfgp/elv014. [PubMed] [CrossRef] [Google Scholar]
Escaramís G., Docampo E., Rabionet R. Десятилетие структурных вариантов: описание, история и методы обнаружения структурных изменений. Краткий. Функц. Геном. 2015;14:305–314. doi: 10.1093/bfgp/elv014. [PubMed] [CrossRef] [Google Scholar]
109. Pirooznia M., Goes F., Zandi P.P. Полногеномный анализ CNV: достижения в вычислительных подходах. Фронт. Жене. 2015;6:1–9. doi: 10.3389/fgene.2015.00138. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
110. Куэйл М.А., Смит М., Коупленд П., Отто Т.Д., Харрис С.Р., Коннор Т.Р., Бертони А., Свердлоу Х.П., Гу Ю.А. рассказ о трех платформах секвенирования NGS. БМС Геном. 2012; 13:1–13. дои: 10.1186/1471-2164-13-341. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
111. Chowell D., Napier J., Gupta R., Anderson K.S., Maley C.C., Wilson Sayres M.A. Моделирование субклональной эволюции популяций раковых клеток. Рак Рез. 2018; 78: 830–839. doi: 10.1158/0008-5472.CAN-17-1229. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
112. Stratton M.R., Campbell P.J., Futreal P.A. Геном рака. Природа. 2009; 458: 719–724. doi: 10.1038/nature07943. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Stratton M.R., Campbell P.J., Futreal P.A. Геном рака. Природа. 2009; 458: 719–724. doi: 10.1038/nature07943. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
113. Gaffney E.F., Riegman P.H., Grizzle W.E., Watson P.H. Факторы, которые стимулируют более широкое использование ткани FFPE в фундаментальных и трансляционных исследованиях рака. Биотех. гистохим. 2018;93: 373–386. doi: 10.1080/10520295.2018.1446101. [PubMed] [CrossRef] [Google Scholar]
114. Reumers J., Rijk P.D., Zhao H., Liekens A., Smeets D., Cleary J., Loo P.V., Bossche M.V.D., Catthor K., Sabbe B. , и другие. Оптимизированная фильтрация снижает частоту ошибок при обнаружении геномных вариантов с помощью секвенирования с коротким считыванием. Нац. Биотехнолог. 2012; 30:61–68. doi: 10.1038/nbt.2053. [PubMed] [CrossRef] [Google Scholar]
Секвенирование нового поколения в клинике: готовы ли мы?
- Опубликовано:
- Leslie G. Biesecker 1 ,
- Wylie Burke 2 ,
- Isaac Kohane 3 ,
- Sharon E. Plon 4 &
- …
- Ron Zimmern 5
Природа Обзоры Генетика том 13 , страницы 818–824 (2012 г.)Процитировать эту статью
12 тыс. обращений
97 цитирований
31 Альтметрический
Сведения о показателях
Субъекты
- Генетика болезней
- Этика
- Генетическое тестирование
- Секвенирование следующего поколения
Abstract
Мы вступаем в эру, когда стоимость клинических полногеномных тестов и тестов целевого секвенирования больше не является препятствием для их применения. Однако в настоящее время отсутствует инфраструктура для поддержки пациентов и врачей, сталкивающихся с результирующими данными. Здесь мы просим пять экспертов высказать свое мнение о том, следует ли рассматривать клинические данные иначе, чем другие медицинские данные, учитывая потенциальное использование этих тестов, а также о областях, которые необходимо развивать для улучшения результатов лечения пациентов.
Однако в настоящее время отсутствует инфраструктура для поддержки пациентов и врачей, сталкивающихся с результирующими данными. Здесь мы просим пять экспертов высказать свое мнение о том, следует ли рассматривать клинические данные иначе, чем другие медицинские данные, учитывая потенциальное использование этих тестов, а также о областях, которые необходимо развивать для улучшения результатов лечения пациентов.
Отличаются ли клинические геномные данные от данных других медицинских тестов? Должны ли они обрабатываться по-другому?
Это предварительный просмотр содержимого подписки, доступ через ваше учреждение
Соответствующие статьи
Статьи открытого доступа со ссылками на эту статью.
Секвенирование РНК выявляет профили экспрессии circRNA и идентифицирует сигнатуру из четырех circRNA, которая действует как прогностический маркер при плоскоклеточной карциноме пищевода.

- Вэйвэй Ван
- , Ди Чжу
- … Гочжун Цзян
Раковая клетка International Открытый доступ 04 марта 2021 г.
Прогностическое значение сигнатуры из пяти днРНК при плоскоклеточном раке пищевода
- Лань Чжан
- , Пань Ли
- … Гочжун Цзян
Раковая клетка, международный Открытый доступ 10 августа 2020 г.

Опыт поставщиков медицинских услуг в отношении возврата результатов геномного секвенирования: интервью
- Джулия Винн
- , Кэти Льюис
- … Сара Сколлон
Медицинская геномика BMC Открытый доступ 08 мая 2018 г.
Варианты доступа
Подписаться на журнал
Получить полный доступ к журналу на 1 год
99,00 €
всего 8,25 € за номер
Подписаться
Расчет налогов будет завершен во время оформления заказа.
Купить статью
Получите ограниченный по времени или полный доступ к статье на ReadCube.
$32,00
Купить
Все цены указаны без учета стоимости.
Ссылки
Burson, C.M. & Markey, K.R. Вопросы генетического консультирования при прогностическом генетическом тестировании семейных неврологических заболеваний у взрослых. Семинары Педиатр. Нейрол. 8 , 177–186 (2001).
КАС Статья Google ученый
Эванс, Дж. П. и Ротшильд, Б. Б. Возврат результатов: не так уж сложно? Жен. Мед. 14 , 358–360 (2012).
Артикул Google ученый
Бизекер, Л. Г. Возможности и проблемы интеграции массового параллельного геномного секвенирования в клиническую практику: уроки проекта ClinSeq. Жен.
 Мед. 14 , 393–398 (2012).
Мед. 14 , 393–398 (2012).Артикул Google ученый
Кохане, И. С., Хсинг, М. и Конг, С. В. Таксономизация, определение размеров и преодоление случайностей. Жен. Мед. 14 , 399–404 (2012).
КАС Статья Google ученый
Медицинский институт Национальной академии. Интеграция крупномасштабной геномной информации в клиническую практику: резюме семинара (издательство национальных академий, 2012 г.).
Mennuti, M. T. Генетический скрининг в охране репродуктивного здоровья. клин. Обст. Гинекол. 51 , 3–23 (2008).
Артикул Google ученый
Goldman, J. S. et al. Генетическое консультирование и тестирование на болезнь Альцгеймера: совместные практические рекомендации Американского колледжа медицинской генетики и Национального общества консультантов-генетиков.
 Жен. Мед. 13 , 597–605 (2011).
Жен. Мед. 13 , 597–605 (2011).Артикул Google ученый
Берк В. Генетическое тестирование. Новый англ. Дж. Мед. 347 , 1867–1875 (2002).
КАС Статья Google ученый
Патай, Б. А. и Тополь, Э. Дж. Неудовлетворенная потребность в образовании в области геномной медицины. утра. Дж. Мед. 125 , 5–6 (2012).
Артикул Google ученый
Сифри, Р. и др. Использование тестирования на предрасположенность к раку среди врачей первичной медико-санитарной помощи. клин. Жене. 64 , 355–360 (2003).
КАС Статья Google ученый
Wideroff, L. et al. Использование врачом генетического тестирования на предрасположенность к раку: результаты национального исследования.
 Рак Эпидемиол. Биомаркеры Пред. 12 , 295–303 (2003).
Рак Эпидемиол. Биомаркеры Пред. 12 , 295–303 (2003).ПабМед Google ученый
Шойнер, М. и др. Готовы ли электронные медицинские карты для геномной медицины? Жен. Мед. 11 , 510–517 (2009).
Артикул Google ученый
Тверски А. и Канеман Д. Суждение в условиях неопределенности: эвристика и предубеждения. Наука 185 , 1124–1131 (1974).
КАС Статья Google ученый
Соловиц П. и Паукер С. Г. Категориальные и вероятностные рассуждения в медицинской диагностике. Артиф. Интеллект Мед. 11 , 115–144 (1978).
Артикул Google ученый
Лин, З., Альтман, Р. Б. и Оуэн, А. Б. Конфиденциальность в исследованиях генома.
 Наука 313 , 441–442 (2006).
Наука 313 , 441–442 (2006).КАС Статья Google ученый
Поттер, Н. Т., Спектор, Э. Б. и Прайор, Т. В. Технические стандарты и рекомендации по тестированию на болезнь Хантингтона. Жен. Мед. 6 , 61–65 (2004).
Артикул Google ученый
Johnston, J.J. et al. Вторичные варианты у лиц, подвергающихся секвенированию экзома: скрининг 572 человек выявил мутации с высокой пенетрантностью в генах предрасположенности к раку. 903:03 утра. Дж. Хам. Жене. 91 , 97–108 (2012).
КАС Статья Google ученый
Райли, Б.Д. и др. Основные элементы оценки риска генетического рака, консультирования и тестирования: обновленные рекомендации Национального общества консультантов-генетиков. Ж. Жене. Счетчики 21 , 151–161 (2012).

Артикул Google ученый
Plon, S. E. et al. Генетическое тестирование и рекомендации врачей по управлению риском рака для родственников из группы риска. Жен. Мед. 13 , 148–154 (2011).
Артикул Google ученый
Голдгар, Д. Э., Истон, Д. Ф., Кэннон-Олбрайт, Л. А. и Сколник, М. Х. Систематическая популяционная оценка риска рака у родственников первой степени родства раковых пробандов. J. Natl Cancer Inst. 86 , 1600–1608 (1994).
КАС Статья Google ученый
Bisecker, L.G. et al. Проект ClinSeq: пилотирование крупномасштабного секвенирования генома для исследований в области геномной медицины. Рез. генома. 19 , 1665–1674 (2009).
КАС Статья Google ученый
Лессер, К.
 С., Люси, К.Р., Эгенер, Б., Брэддок, С.Х., Линас, С.Л. и Левинсон, В. Поведенческий и системный взгляд на профессионализм. JAMA 304 , 2732–2737 (2010).
С., Люси, К.Р., Эгенер, Б., Брэддок, С.Х., Линас, С.Л. и Левинсон, В. Поведенческий и системный взгляд на профессионализм. JAMA 304 , 2732–2737 (2010).КАС Статья Google ученый
Кохан И.С., Масис Д.Р. и Альтман Р.Б. Инциденталом: угроза геномной медицине. JAMA 296 , 212–215 (2006).
КАС Статья Google ученый
Roberts, N.J. et al. Прогностическая способность персонального секвенирования генома. Науч. Перевод Мед. 4 , 133ra58 (2012).
Артикул Google ученый
Gahl, W. A. et al. Программа национальных институтов здравоохранения по недиагностированным заболеваниям: взгляд на редкие заболевания. Жен. Мед. 14 , 51–59 (2012).
КАС Статья Google ученый
Максмен, А.
 Секвенирование экзома для расшифровки редких заболеваний. Cell 144 , 635–637 (2011).
Секвенирование экзома для расшифровки редких заболеваний. Cell 144 , 635–637 (2011).КАС Статья Google ученый
Mayer, A. N. et al. Своевременное появление геномной медицины. Жен. Мед. 13 , 195–196 (2011).
Артикул Google ученый
Берг, Дж. С. и др. Информатический подход к анализу инциденталома. Жен. Мед. , 27 июля 2012 г. (doi:10.1038/gim.2012.112).
Hicks, J.K. et al. Управляемая врачом автоматизированная система для интеграции фармакогенетической интерпретации в электронную медицинскую карту. клин. Фармакол. тер. , 19 сентября 2012 г. (doi: 10.1038/clpt.2012.140).
Оверби, С. Л., Тарчи-Хорнох, П., Хоат, Дж. И., Калет, И. Дж. и Винстра, Д. Л. Возможность включения геномных знаний в электронные медицинские записи для поддержки фармакогеномных клинических решений.
 BMC Биоинформатика 11 (Прил. 9), S10 (2010).
BMC Биоинформатика 11 (Прил. 9), S10 (2010).Артикул Google ученый
Гуттмахер А. Э., Дженкинс Дж. и Ульманн В. Р. Геномная медицина: кто будет ее практиковать? Призыв к распростертым объятиям. утра. Дж. Мед. Жене. 106 , 216–222 (2001).
КАС Статья Google ученый
Millot, G. A. et al. Руководство по функциональному анализу BRCA1 варианта неопределенной значимости. Гул. Мутат. , 16 июля 2012 г. (doi: 10.1002/humu.22150).
Райт, К. и др. Следующие шаги в последовательности: последствия полногеномного секвенирования для здоровья в Великобритании Ch. 12 153–160 (Фонд PHG, 2011).
Google ученый
Райт, К. и др. Следующие шаги в последовательности: последствия полногеномного секвенирования для здоровья в Великобритании Гл.
 4 33–44 (Фонд PHG, 2011).
4 33–44 (Фонд PHG, 2011).Google ученый
Farley, T. A. Реформирование здравоохранения или реформирование здоровья? утра. Дж. Общественное здравоохранение. 99 , 588–590 (2009).
Артикул Google ученый
Комитет по разработке новой таксономии болезней; Национальный исследовательский совет. На пути к точной медицине: создание сети знаний для биомедицинских исследований и новой таксономии болезней (издательство национальных академий, 2011 г.).
Qi, Q. et al. Сахаросодержащие напитки и генетический риск ожирения. Н. англ. Дж. Мед. , 21 сентября 2012 г. (doi: 10.1056/NEJMoa1203039).
Ссылки на скачивание
Благодарности
L.G.B. и программа ClinSeq поддерживаются Программой внутренних исследований Национального института исследований генома человека США. С.Э.П. подтверждает предоставление 1 U01 HG006485. Р.З. хотел бы поблагодарить M. Kroese и A. Hall, оба из PHG Foundation, за полезное обсуждение. Высказанные мнения являются собственными авторами и не обязательно отражают точку зрения учреждений, с которыми они связаны.
С.Э.П. подтверждает предоставление 1 U01 HG006485. Р.З. хотел бы поблагодарить M. Kroese и A. Hall, оба из PHG Foundation, за полезное обсуждение. Высказанные мнения являются собственными авторами и не обязательно отражают точку зрения учреждений, с которыми они связаны.
Информация об авторе
Авторы и организации
Лесли Г. Бизекер работает в Отделе исследований генетических заболеваний, Национальный исследовательский институт генома человека, 49 Convent Drive, Room 4A56, Бетесда, Мэриленд 20892, США.,
Лесли Г. Бизекер
Уайли Берк работает на кафедре биоэтики и гуманитарных наук Вашингтонского университета, BOX 357120, 1959 NE Pacific, Room A204, Сиэтл, Вашингтон 98195–7120, США.,
Wylie Burke
Исаак Кохейн находится в Бостонской детской больнице, 300 Longwood Avenue, Enders-6, Boston Massachusetts 02115, USA.,
Исаак Кохан
Шэрон Э.
 Плон работает в отделениях педиатрии, гематологии-онкологии и молекулярной генетики человека в Центре секвенирования генома человека Медицинского колледжа Бейлора, Клиники генетики рака Бейлора, Детской больницы Техаса, Хьюстон, Техас 77030, США.,
Плон работает в отделениях педиатрии, гематологии-онкологии и молекулярной генетики человека в Центре секвенирования генома человека Медицинского колледжа Бейлора, Клиники генетики рака Бейлора, Детской больницы Техаса, Хьюстон, Техас 77030, США.,Шарон Э. Плон
Рон Циммерн из Фонда PHG, 2 Worts Causeway, Cambridge CB1 8RN, UK.,
Рон Циммерн
Авторы
- Лесли Г. Бизекер
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Wylie Burke
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
- Isaac Kohane
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Sharon E. Plon
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Ron Zimmern
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Авторы переписки
Переписка с
Лесли Г. Бизекер, Уайли Берк, Исаак Кохейн, Шэрон Э. Плон или Рон Циммерн.
Бизекер, Уайли Берк, Исаак Кохейн, Шэрон Э. Плон или Рон Циммерн.
Декларации этики
Конкурирующие интересы
Лесли Г. Бизекер является безвозмездным консультантом и сотрудником Illumina Corporation. Уайли Берк, Исаак Кохейн, Шэрон Э. Плон и Рон Циммерн заявляют об отсутствии конкурирующих финансовых интересов.
Ссылки по теме
ДОПОЛНИТЕЛЬНАЯ ИНФОРМАЦИЯ
Домашняя страница Лесли Г. Бизекера
Домашняя страница Уайли Берк
Домашняя страница Исаака Кохана
Домашняя страница Шэрон Э. Плон
Домашняя страница Рона Циммерна
Оценка генетического риска рака и консультирование (PDQ) — Национальный институт рака
Исследовательское клиническое секвенирование — Национальный исследовательский институт генома человека США
Права и разрешения
Перепечатка и разрешения
Об этой статье
Эта статья цитируется
Секвенирование РНК выявляет профили экспрессии circRNA и идентифицирует сигнатуру из четырех circRNA, которая действует как прогностический маркер при плоскоклеточной карциноме пищевода.

- Вэйвэй Ван
- Ди Чжу
- Гуочжун Цзян
Cancer Cell International (2021)
Прогностическое значение сигнатуры из пяти днРНК при плоскоклеточном раке пищевода
- Лань Чжан
- Пан Ли
- Гуочжун Цзян
Cancer Cell International (2020)
Лабораторный подход к гемолитической анемии
- Ману Джамвал
- Прашант Шарма
- Рина Дас
Индийский журнал педиатрии (2020)
Гены, кодирующие белок, в сочетании с длинной некодирующей РНК как новая модель молекулярного стадирования транскриптома для прогнозирования выживаемости пациентов с плоскоклеточным раком пищевода
- Цзинь-Чэн Го
- Ян Ву
- Ли-Ян Сюй
Сообщения о раке (2018)
Опыт поставщиков медицинских услуг в отношении возврата результатов геномного секвенирования: интервью
- Джулия Винн
- Кэти Льюис
- Сара Сколлон
BMC Medical Genomics (2018)
Секвенирование нового поколения в клинике
- Опубликовано:
- Джейсон И. Парк 1 ,
- Ларри Дж. Крика 2 и
- Паоло Фортина 3
Природные биотехнологии том 31 , страницы 990–992 (2013 г.)Процитировать эту статью
8497 Доступ
30 цитирований
8 Альтметрический
Сведения о показателях
Субъекты
- Геномика рака
- Политика здравоохранения
- Секвенирование следующего поколения
Пулы клеточных линий, несущих множество известных мутаций, используются для проверки эффективности диагностического теста на рак, основанного на секвенировании нового поколения.
По мере того, как секвенирование опухолевых клеток нового поколения (NGS) становится все более изощренным, оно, вероятно, будет использоваться во всех аспектах лечения рака, от диагностического тестирования до лечения и разработки лекарств. В этом выпуске Frampton и др. . 1 описывают комплексный анализ NGS, применимый к клиническим образцам, который выявляет однонуклеотидные замены, вариации числа копий и фокальные амплификации в 287 генах, связанных с раком, события слияния с участием 19 часто перестраиваемых генов и 3549однонуклеотидные полиморфизмы в других местах генома — все в рамках одного цикла секвенирования. Проверка аналитической эффективности такого теста является сложной задачей, поскольку он анализирует очень много позиций нуклеотидов в геноме (всего ~ 1,5 Мб). Поэтому авторы используют 53 клеточные линии для создания эталонных материалов для оценки чувствительности и специфичности обнаружения вариантов. Наконец, они применяют свой тест к более чем 2000 клинических случаев. Общий подход исследования, включая дизайн анализа, создание эталонных материалов и проверку, служит моделью для разработки будущих клинических тестов NGS.
Общий подход исследования, включая дизайн анализа, создание эталонных материалов и проверку, служит моделью для разработки будущих клинических тестов NGS.
Это предварительный просмотр содержимого подписки, доступ через ваше учреждение
Соответствующие статьи
Статьи открытого доступа со ссылками на эту статью.
Сетевой анализ факторов транскрипции, основанный на РНК-seq одиночных клеток, показывает, что трихостатин-а меняет резистентность к доцетакселу при раке предстательной железы.
- Патрисия М. Шнепп
- , Акила Ахмед
- … Эван Т. Келлер
Рак BMC Открытый доступ 08 декабря 2021 г.

Сравнение производительности выбранных вызывающих вариантов с использованием синтетических данных и сегментации генома
- Сяопэн Бянь
- , Бин Чжу
- … Дауд Мирзаман
Биоинформатика BMC Открытый доступ 19 ноября 2018 г.
ToTem: инструмент для оптимизации пайплайна вариантов вызова
- Никола Том
- , Ондрей Том
- … Сарка Посписилова
Биоинформатика BMC Открытый доступ 26 июня 2018 г.

Варианты доступа
Подписаться на журнал
Получить полный доступ к журналу на 1 год
99,00 €
всего 8,25 € за номер
Подписаться
Расчет налогов будет завершен во время оформления заказа.
Купить статью
Получите ограниченный по времени или полный доступ к статье на ReadCube.
$32,00
Купить
Все цены указаны без учета стоимости.
Рис. 1: Протокол клинического секвенирования и проверка.Ссылки
Frampton, G.M. и другие. Нац. Биотехнолог. 31 , 1023–1031 (2013).
КАС Статья Google ученый
Конти, Р.М. и другие. Дж. Клин. Онкол. 31 , 1134–1139 (2013).

КАС Статья Google ученый
Гаргис А.С. и другие. Нац. Биотехнолог. 30 , 1033–1036 (2012).
КАС Статья Google ученый
Rehm, HL et al. Жен. Мед. 15 , 733–747 (2013).
Артикул Google ученый
Программа аккредитации лабораторий CAP. Контрольный список молекулярной патологии http://www.cap.org/apps/docs/laboratory_accreditation/checklists/new/молекулярное_патология_checklist.pdf (Колледж американских патологоанатомов, 2013 г.).
Bose, R. et al. Рак Дисков. 3 , 224–237 (2013).
КАС Статья Google ученый
Кржижановска М.К. Дж. Клин. Онкол. 31 , 1125–1127 (2013).

КАС Статья Google ученый
Альбертс, С.Р. и другие. Дж. Ам. Мед. доц. 307 , 1383–1393 (2012).
КАС Статья Google ученый
Garber, K. J. Natl. Рак инст. 103 , 84–86 (2011).
Артикул Google ученый
Simon, R. & Roychowdhury, S. Nat. Преподобный Друг Дисков. 12 , 358–369 (2013).
КАС Статья Google ученый
Скачать ссылки
Информация об авторе
Авторы и организации
Джейсон Ю. Парк работает в отделении патологии Юго-Западного медицинского центра Техасского университета и Детского медицинского центра, Даллас, Техас, США, и Юджина. Центр человеческого роста и развития Макдермотта, Юго-западный медицинский центр Техасского университета, Даллас, Техас, США.
 ,
,Jason Y Park
Ларри Дж. Крика работает в отделении патологии и лабораторной медицины Медицинского центра Пенсильванского университета, Филадельфия, Пенсильвания, США.
Ларри Дж. Крика
Кафедра биологии рака, Паоло Фортина работает в Лаборатории геномики рака, Онкологический центр Киммела, Университет Томаса Джефферсона, Медицинский колледж Джефферсона, Филадельфия, Пенсильвания, США.,
Paolo Fortina
Авторы
- Jason Y Park
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
- Larry J Kricka
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
- Paolo Fortina
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Автор, ответственный за корреспонденцию
Джейсон И Парк.
Заявление об этике
Конкурирующие интересы
Авторы не заявляют об отсутствии конкурирующих финансовых интересов.
Права и разрешения
Перепечатка и разрешения
Об этой статье
Дополнительная литература
Сетевой анализ факторов транскрипции, основанный на РНК-seq одиночных клеток, показывает, что трихостатин-а меняет резистентность к доцетакселу при раке предстательной железы.
- Патрисия М. Шнепп
- Акила Ахмед
- Эван Т. Келлер
Рак BMC (2021)
Сравнение производительности выбранных вызывающих вариантов с использованием синтетических данных и сегментации генома
- Сяопэн Бянь
- Бин Чжу
- Дауд Мирзаман
Биоинформатика BMC (2018)
ToTem: инструмент для оптимизации пайплайна вариантов вызова
- Никола Том
- Ондрей Том
- Сарка Посписилова
Биоинформатика BMC (2018)
Оценка инструментов вызова вариантов для несовпадающих данных секвенирования следующего поколения
- Сара Сандманн
- Аник О.




 Мед. 14 , 393–398 (2012).
Мед. 14 , 393–398 (2012). Жен. Мед. 13 , 597–605 (2011).
Жен. Мед. 13 , 597–605 (2011). Рак Эпидемиол. Биомаркеры Пред. 12 , 295–303 (2003).
Рак Эпидемиол. Биомаркеры Пред. 12 , 295–303 (2003). Наука 313 , 441–442 (2006).
Наука 313 , 441–442 (2006).
 С., Люси, К.Р., Эгенер, Б., Брэддок, С.Х., Линас, С.Л. и Левинсон, В. Поведенческий и системный взгляд на профессионализм. JAMA 304 , 2732–2737 (2010).
С., Люси, К.Р., Эгенер, Б., Брэддок, С.Х., Линас, С.Л. и Левинсон, В. Поведенческий и системный взгляд на профессионализм. JAMA 304 , 2732–2737 (2010). Секвенирование экзома для расшифровки редких заболеваний. Cell 144 , 635–637 (2011).
Секвенирование экзома для расшифровки редких заболеваний. Cell 144 , 635–637 (2011). BMC Биоинформатика 11 (Прил. 9), S10 (2010).
BMC Биоинформатика 11 (Прил. 9), S10 (2010). 4 33–44 (Фонд PHG, 2011).
4 33–44 (Фонд PHG, 2011). Плон работает в отделениях педиатрии, гематологии-онкологии и молекулярной генетики человека в Центре секвенирования генома человека Медицинского колледжа Бейлора, Клиники генетики рака Бейлора, Детской больницы Техаса, Хьюстон, Техас 77030, США.,
Плон работает в отделениях педиатрии, гематологии-онкологии и молекулярной генетики человека в Центре секвенирования генома человека Медицинского колледжа Бейлора, Клиники генетики рака Бейлора, Детской больницы Техаса, Хьюстон, Техас 77030, США.,



 ,
,